FAQ(2020/1/10更新)
前提:AVIZO/amira, Predictが準備されている事。
|
|
|
追加モジュール |
追加日 |
||
|
Q1 |
要 |
|
|||
|
Q2 |
要 |
|
|||
|
Q3 |
要 |
|
|||
|
Q4 |
|
|
|||
|
Q5 |
|
|
|||
|
Q6 |
要 |
|
|||
|
Q7 |
|
|
|||
|
Q8 |
【連結性評価その1】表面にできた凹くぼみ、内在する空洞、貫通する穴を区別できますか? |
|
|
||
|
Q9 |
【連結性評価その2】第二相の様々な形状のオイラー標数、種数の評価はどうしますか? |
|
|
||
|
Q10 |
|
2017/8/29 |
|||
|
|
|
||||
|
|
【追加モジュールについて】 *AVIZO/amiraの拡張オプション 問い合わせ先: Avizo/amiraのwatershed機能解析には拡張オプションが必要です。 Avizo “Fire 6〜8.1” Avizo9.0以降 Amira6 “XImagaPAQ拡張機能” |
||||
|
|
|
||||
|
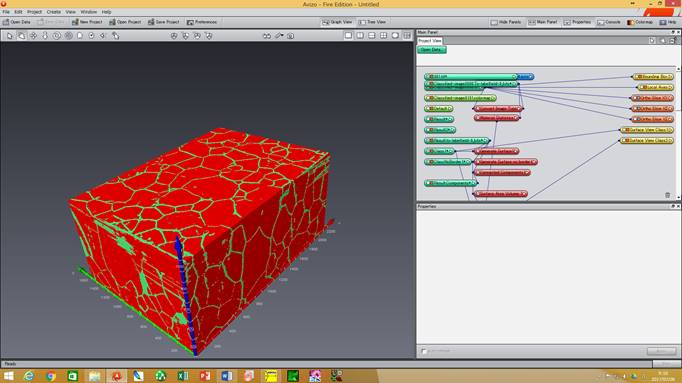
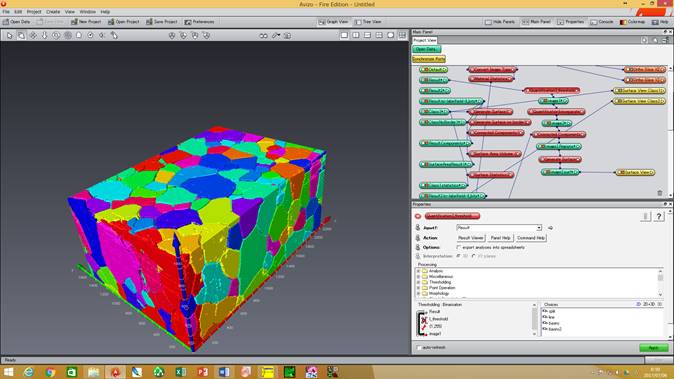
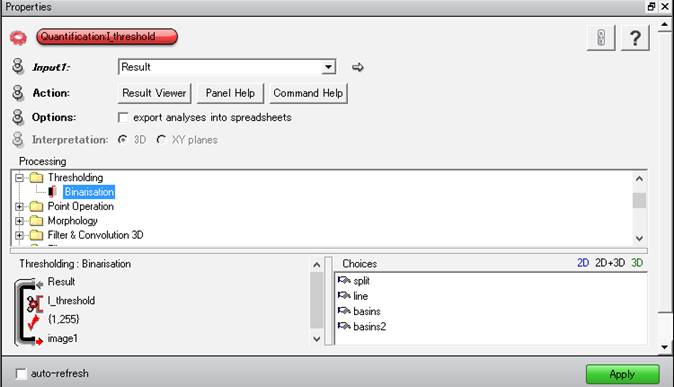
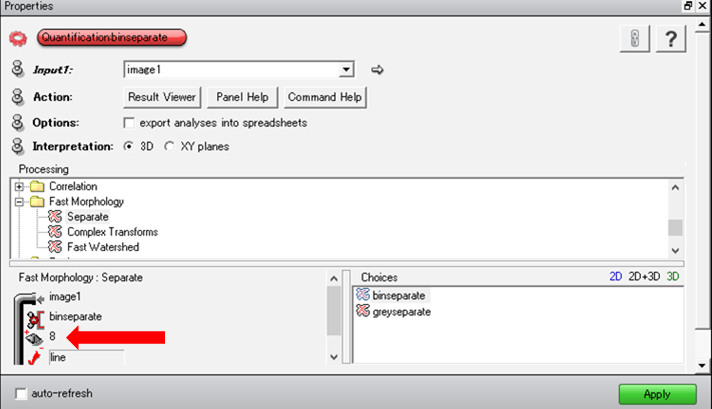
*AVIZO/amiraの拡張オプションが必要です。 MIPHAによりおおよその可視化は可能ですが、一部粒界が分断されているところが残ります。その追加処置として、Avizo/amiraのwatershed機能を使います。この機能は以下の拡張オプションが必要です。 AvizoFire 6〜8.1 Avizo9.0以降 Amira6 XImagaPAQ拡張機能 *Avizo7とAvizo9またはAmira6でモジュール名が異なりますのでご注意下さい。 【AvizoFire6〜8.1の場合】 (1)粒と粒界のラベルデータに対して、I_thrsholdをつなぎ、1-255としてApplyする。これで、Watershed処理に使う粒界が黒、粒が白の画像データが作成されます(画像そのものは同じですが、形式が違います)。 (2)「Result」にQuantification Tool/binseparateをつなぎます。パラメータの数値を小さくすると分割が細かくなります。この場合のWatershedは粒界を描き足すイメージです。 (3)Connected Componentでそれぞれの粒を認識させて、生成した***Regions*よりGenerate SurfaceでSurfaceまで作成します。 【Avizo9/amira6の場合】 【Avizo9/amira6 の場合】 (1)「Result-labelfield-8_bits」に Image Processing -> Separating And Filling -> Separate Objects をつなぎます。パラメータの数値を小さくすると分割が細かくなります。この場合の Watershed は粒界を描き足すイメージです。 (2)Image segmentation/Connected Component (Resultsの下矢印を広げて、Label imageにチェックする。できるだけカラー数を増やしたいので、Output typeをLabel field(32bit)を選択する。生成したResult.Regeins*のshared colormapをlabels256.amにする)でそれぞれの粒を認識させて Surface まで作成します。 このwatershed3D像から、任意の粒だけを取り出して3D表示する事も可能です。 (1)全ての粒が表示されているSurface Viewで、BufferのClearボタンを押下し、全ての粒が赤く示された状態で、Removeを押下します。 (2)何も表示されなくなったら、Materialsのコンボボックスの左側のMaterial番号を選択します。 (3)選択中のMaterialが赤く示されますので、確認して、Addボダンを押します。 ・MIPHAで処理
・追加でWatershed処理を実施 Watershed Quantification
I_threshhold
Quantification binseparate(値が小さいほど粒を分割)
Connected component(Label imageのチェックが必要。Avizo9/amira6の場合は⇩Resultをクリックして詳細設定画面を表示して作業する。)
|
|||||
|
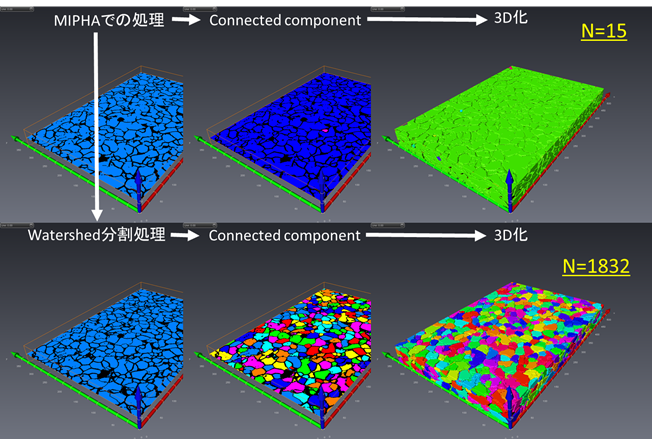
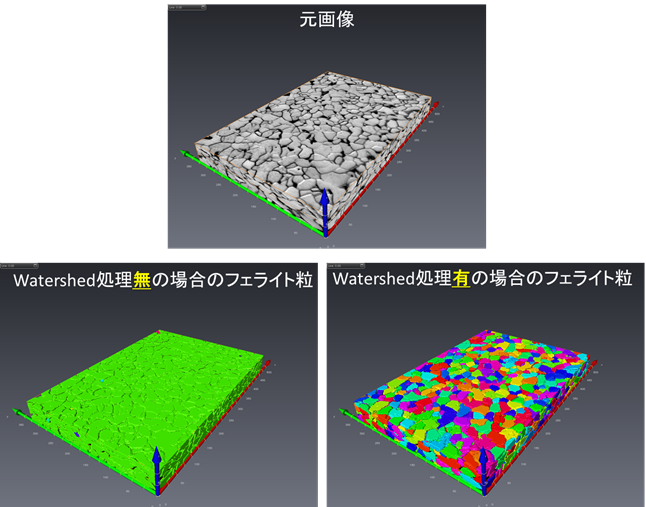
複相組織(例えばDP鋼)中の(フェライト)母相の結晶粒の可視化はMIPHAで可能ですか? *AVIZO/amiraの拡張オプションが必要です。 A1の例と同様に、MIIPHAによる一次処理でおおよその母相の結晶粒形態は把握できますが、粒界が鮮明でないため結晶粒が連結しているように3D可視化され、その結果、結晶粒の個数が非常に小さな値になります(例では15個)。平均結晶粒径を求める際にはこれは大きな問題になります。そこで、該当する相のラベル像に対して、A1と同様にwatershed処理を行います。粒は分割され、元画像の結晶粒に対応したラベリング像が得られます。結晶粒の個数も大幅に増えます(1832個)。粒界が不鮮明な時、正確な粒の数を求める際にはこのように注意が必要です。この機能は以下の拡張オプションが必要です。 AvizoFire 6〜8.1 Avizo9.0以降 Amira6 XImagaPAQ拡張機能
Watershed処理を実施
元画像とWatershed処理の有無によるラベリング結果の比較
|
|||||
|
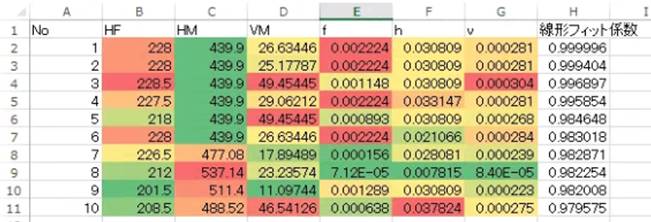
*二番目の方法はニューラルネットワーク解析ソフトPredict及び追加モジュールパック”MAX”が必要です。 二つの方法を提案します。 1つは、逆解析結果のcsvデータを、excelの条件付き書式➡カラースケール表示し、特徴をつかむ方法です。
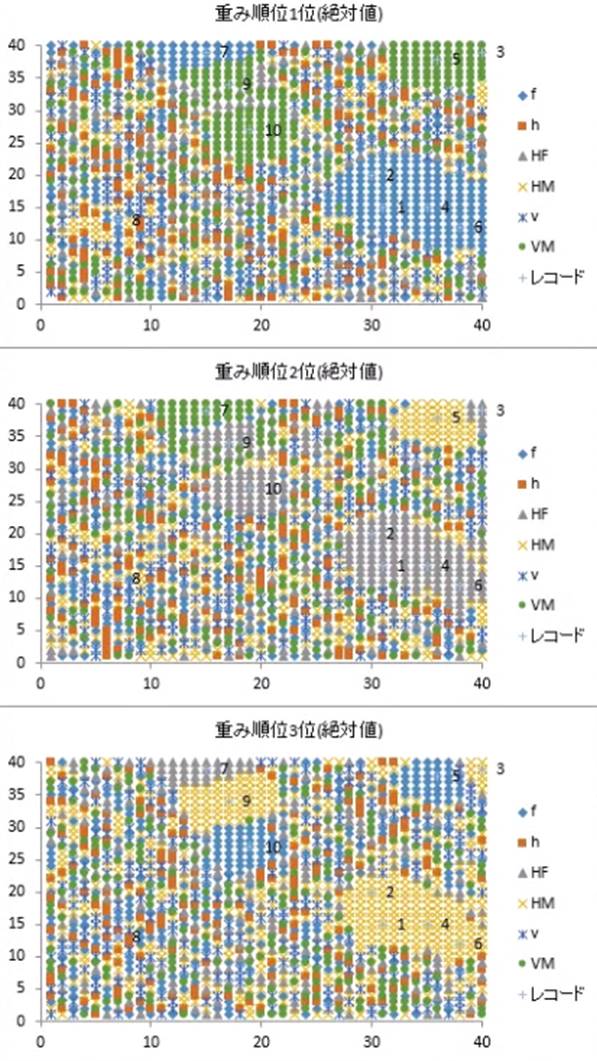
もう一つはの方法は、ニューラルネットワーク解析ソフト“Predict”の自己組織化マップ(SOM)を使う方法です。Predictと追加オプションを使うと、提案された10候補の組織の類似度とその特徴(f,h,HFなど)が分かる図が表示できます。試料番号が近いほど組織は類似しています。例えば、1.の組織、2.の組織は類似しておりその特徴は以下の通りです。試料9はやや離れた位置にプロットされており、組織の特徴が異なります。
一番目の特徴 二番目の特徴 三番目の特徴 試料1 fの影響が大 HFの影響が大 HMの影響が大 試料5 fの影響が大 HFの影響が大 HMの影響が大 試料9 VMの影響が大 HFの影響が大 HMの影響が大 |
|||||
|
A5 |
Fiji、あるいはTrainable
Weka segmentationは自動更新しないことを推奨します。以前作った識別機が読み込めなくなる可能性があります。 |
||||
|
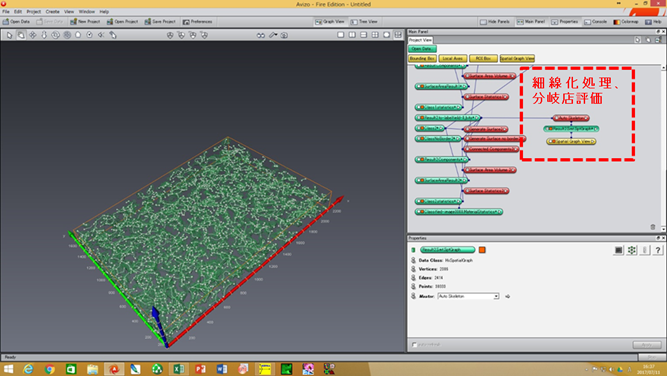
*一部、追加モジュールパック”MAX”が必要です。
次に、Auto skeleton操作によってできた***.SptGraphを右クリックして、Save data Asを選択し、「ファイルの種類」より拡張子.mv3dを選択し、デスクトップに保存します。 追加モジュール(分岐点解析モジュール)を立ち上げて、先ほど保存した.mv3dファイルを選択すると、分岐点解析が実行されます。現在、3,4,5分岐点数の評価が可能です。
|
|||||
|
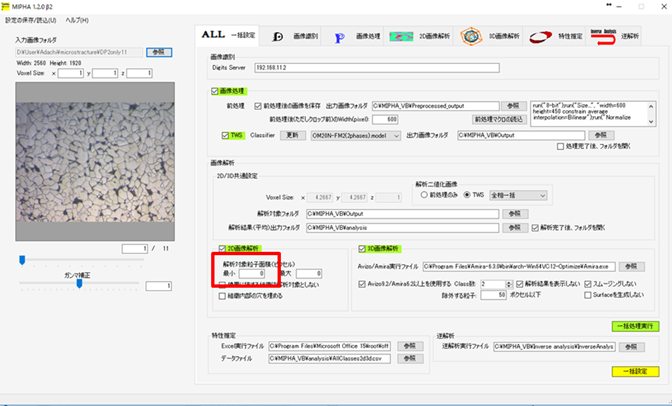
一括実行時に、2D解析結果のcsvファイルが作成されないときがあります。対応策はありますか? (マニュアル記載内容と同じです) 解析対象の粒子数が数万個と非常に多い場合、2D解析結果のcsvファイルの書き込みに長時間を要し(1,2時間かかる場合があります)、Allclasses2d.csvを更新する前に3D定量解析が始まる場合があります。最終結果の更新時間を参考に、正常に2dデータが更新されているかどうかをご確認ください。この対策としては、「2D解析対象の粒子面積(ピクセル)の最小」(下図赤枠)にある値(例えば20)を入力するとこの問題を回避できる場合があります。
|
|||||
|
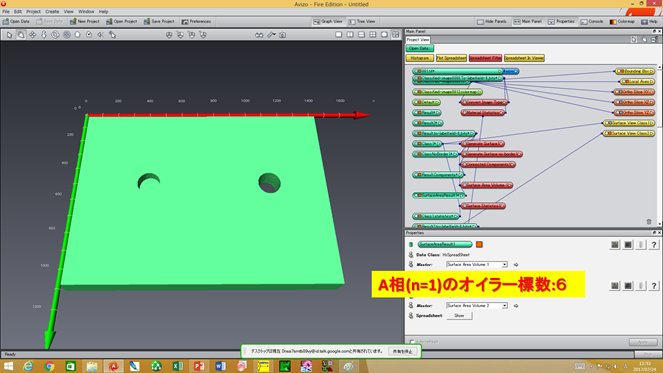
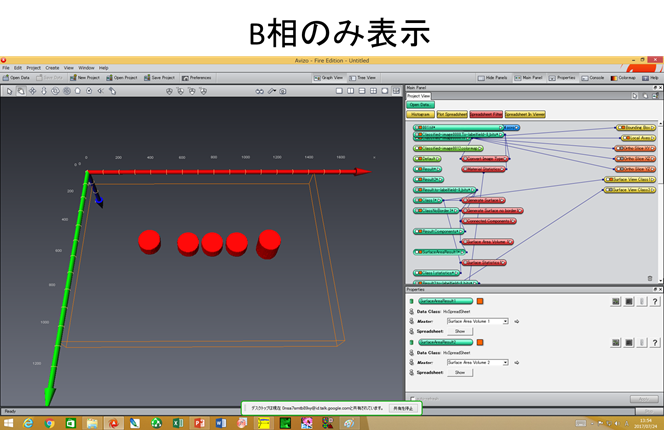
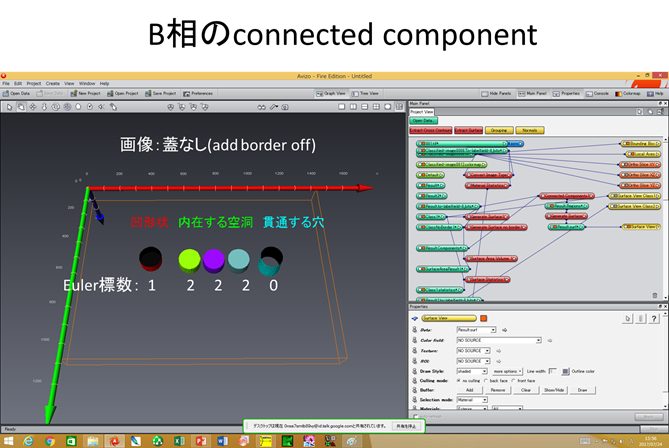
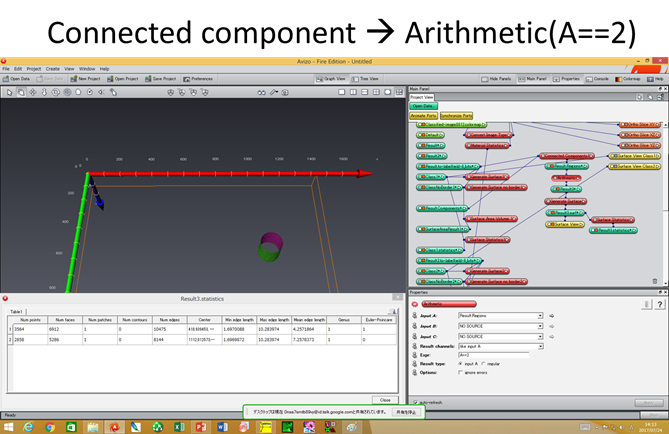
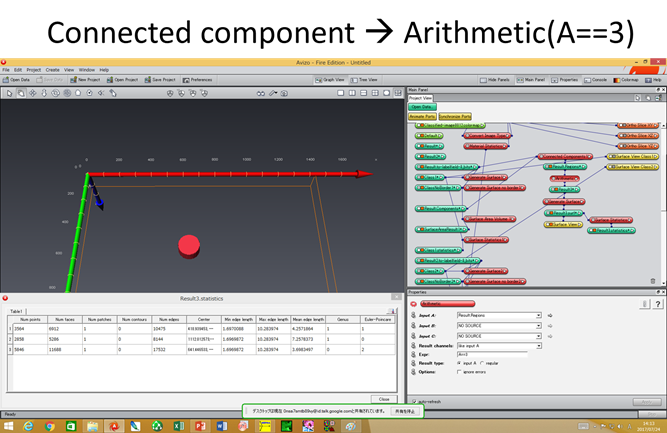
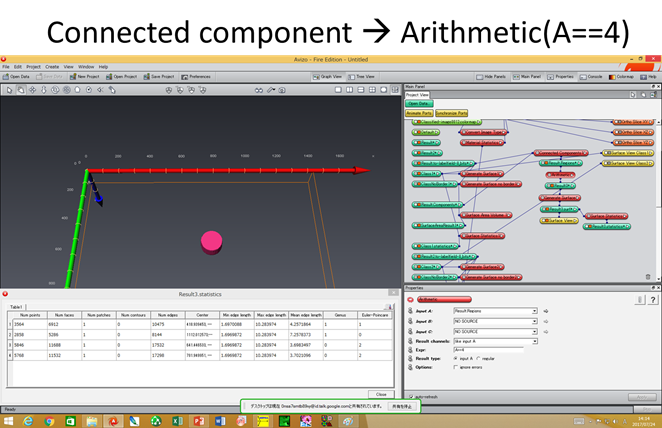
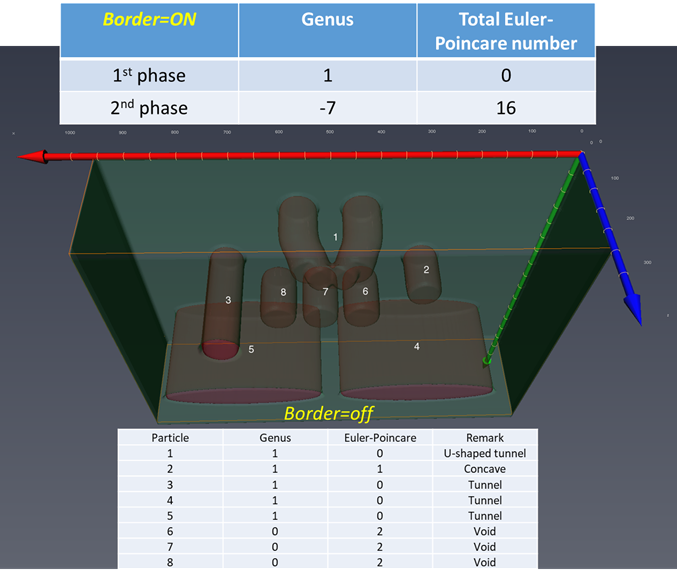
表面にできた凹くぼみ、内在する空洞、貫通する穴を区別できますか? A相の中で、 ・表面にできた凹(=A相中に食い込んだ凸形状のB相) ・内在する空洞(=孤立してA中に存在するB相) ・貫通する穴(A相を貫通して存在するB相) があるとき、それぞれの”B相”のオイラー標数χsurfaceは、 ・表面にできた凹(=A相中に食い込んだ凸形状のB相) Euler数 1 ・内在する空洞(=孤立してA相中に存在するB相) Euler数 2 ・貫通する穴(A相を貫通して存在するB相) Euler数 0 となります。 凹形状や貫通した穴をAVIZO/amiraで描いてオイラー標数を評価するためには、AVIZO/amiraのgenerate surfaceで”add border”のチェックを外します。これにより蓋無しの画像を描いて、上の3つの場合を評価するとそれぞれの形状を区別できます。A相の連結性(=貫通した穴の数)を評価するときには、g=t-(v+b)+1=
|
|||||
|
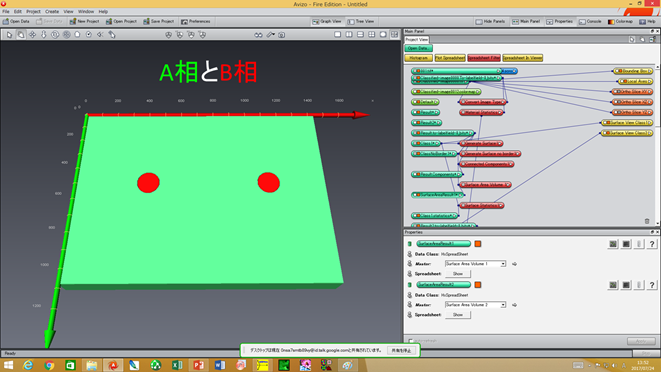
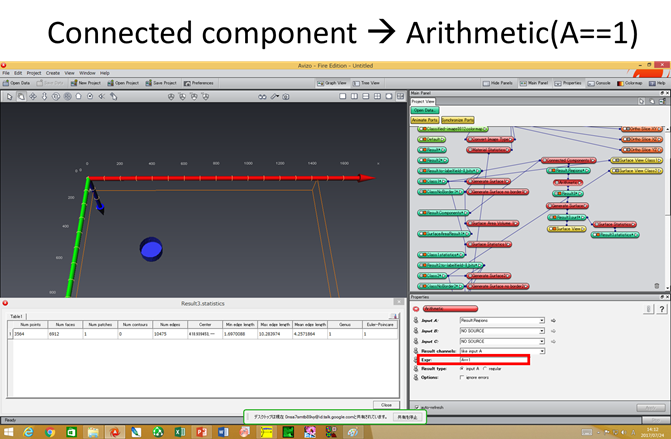
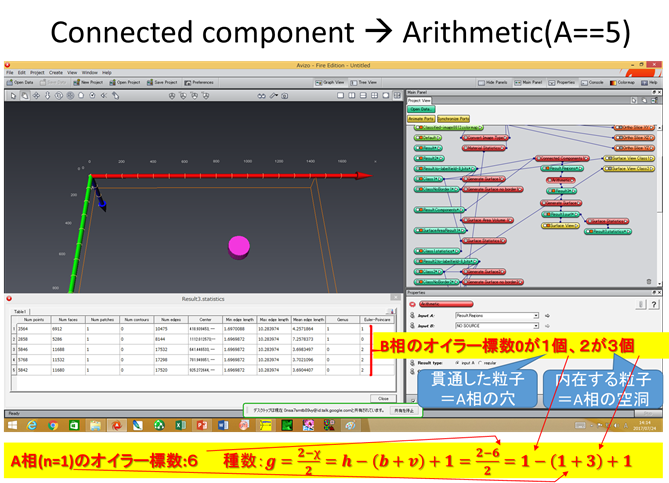
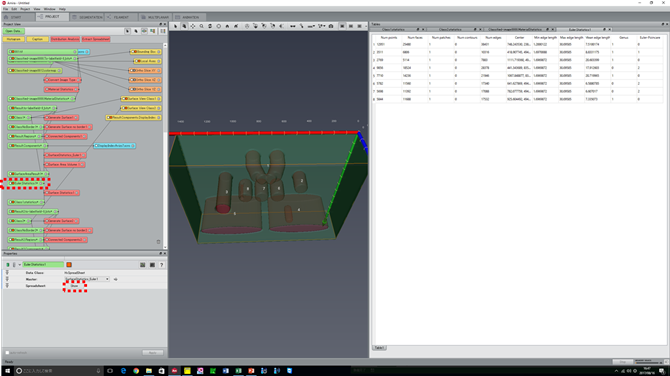
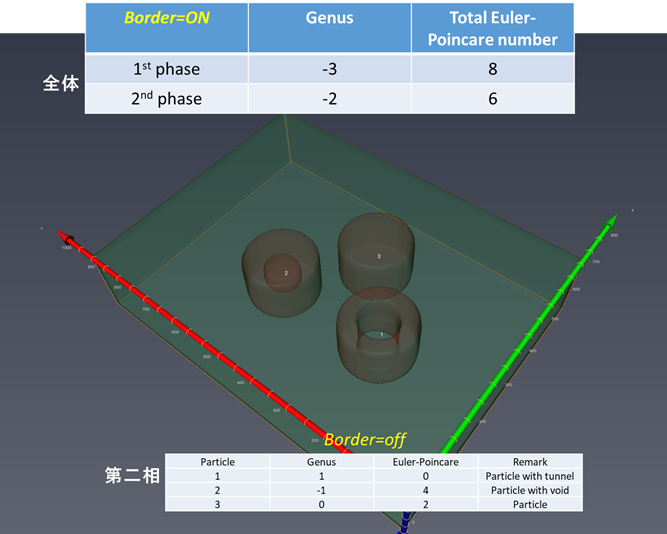
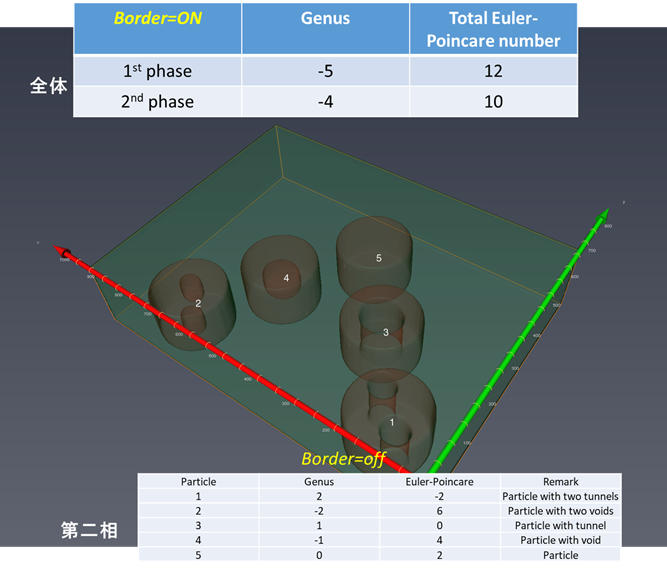
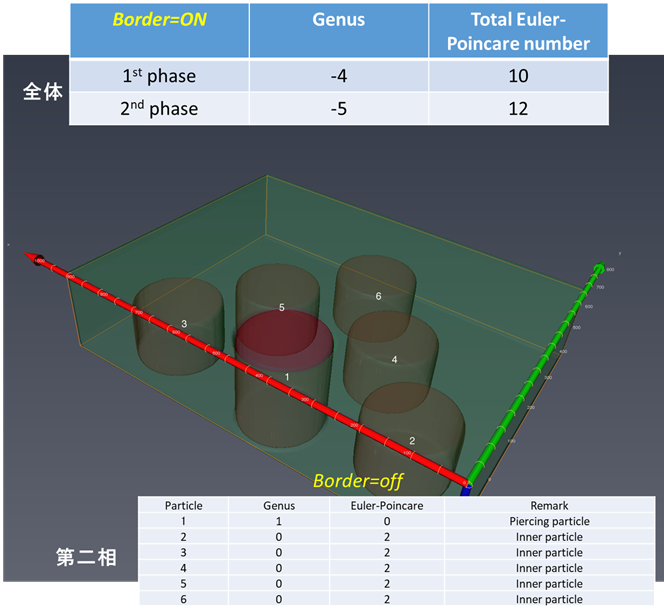
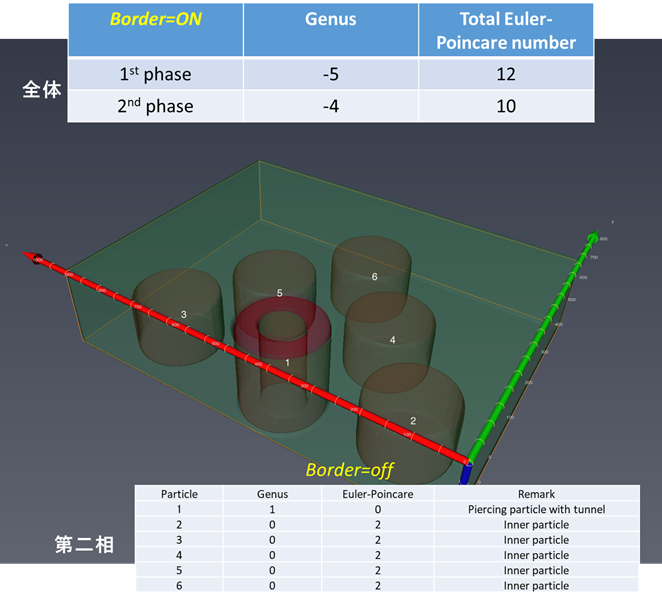
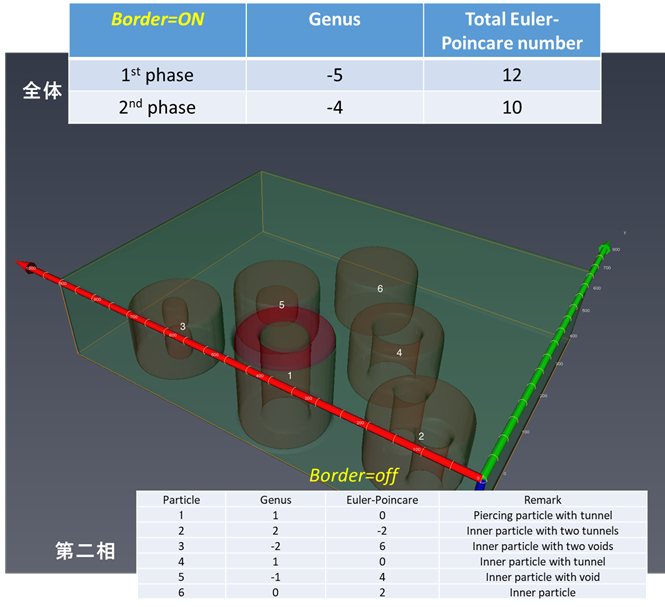
第二相の様々な形状のオイラー標数、種数の評価はどうしますか?2017.8.17ver.1.3.1よりMIPHA(+AVIZO/amira)でこの評価が可能になりました。
上図で、該当する相の”Euler statics”をクリックし、Spreadsheetの”Show”をクリックすると、Genus, Euler-Poincare標数の一覧表が表示されます(CSVでも保存可能です)。 このようにして評価した一例を下に示します。
個々の第二相(母相側からすれば貫通する穴や内在するボイドに相当)の形状をオイラー標数を通じて解析できるとともに、それぞれの形状(オイラー標数χ2=0,2,4 (それぞれ、貫通する第二相粒子、内在する第二相粒子、母相を内側に内側に含む内在する第二相粒子に対応)を有する粒子の数がMIPHA_VBフォルダ内のanalysisフォルダのAllClasses2d3d.csvに一括して集約されます。 例1
例2
例3
例4
例5
|
|||||
|
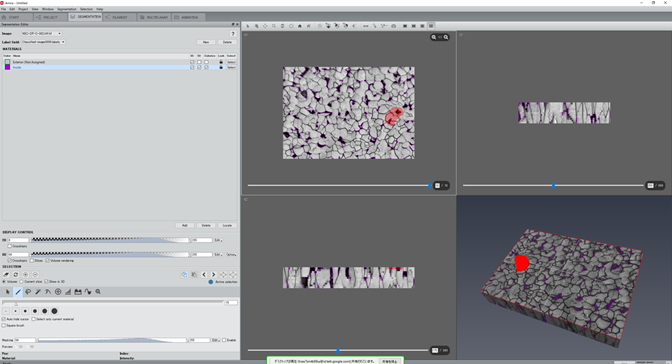
MIPHAで画像処理したラベリング結果の一部を手動で修正したいのですが可能ですか? 可能です。まずAmira/avizoにMIPHAの前処理ホルダより画像を読み込んでください。 次に、outputフォルダよりMIPHAで処理したラベリング像を読み込んでください。そしてthresholdにより対象輝度値の範囲を指定して再ラベリングを行って下さい。この再ラベリング像を再再ラベリングする際に、一番左上のところにあるImageとして前処理画像を指定することにより、組織像と再再ラベリング像を重ね合わせて表示することが可能です。この重ね合わせ像を見ながらラベリングの手動修正を行います。修正後、project画面で修正したラベリング像をもとのラベリング像と入れ替えて保存してください(あるいは他の任意のホルダに保存してください)。 なお、Amira5以降の全バージョン、Avizo9以降の全バージョンでは、Display controlの”Volume rendering”にチェックを入れることにより、ボリュームレンダリング像とラベルデータを重ねて表示することが可能です。
|
|||||